
Protein-Peptid-Docking
Softwaretechnik-Praktikum im Sommersemester 2003
Universität des Saarlandes – Informatik
Informatik Campus des Saarlandes
Campus E9 1 (CISPA)
66123 Saarbrücken
E-mail: zeller @ cs.uni-saarland.de
Telefon: +49 681 302-70970



Beschreibung
Die zunehmende Bedeutung von Peptiden als Medikamente wird sehr deutlich, wenn man im Internet nach neuen Entwicklungen im Bereich der Wirkstoffentwicklung sucht. Peptide werden heute als potentielle Medikamente gegen Krebs, Herz- und Gefäßerkrankungen, Stoffwechselkrankheiten und gegen bakterielle und virale Infektionen gehandelt. Ihre Wirkung entfalten sie, indem sie spezifische Reaktionen mit bestimmten Zielmolekülen tätigen, etwa, indem sie das aktive Zentrum eines vorgegebenen Enzyms blockieren.Im Rahmen dieses Projektes wird ein interaktives Zielmolekül-Peptid-Docking implementiert und getestet werden. Hierzu realisieren wir eine Monte-Carlo-Simulation, die potentielle Zielmolekül-Peptid-Komplexe und deren Bindungsenergie berechnet. Die Simulation wird mit Hilfe unseres Molekül-Viewers MolView visualisiert.
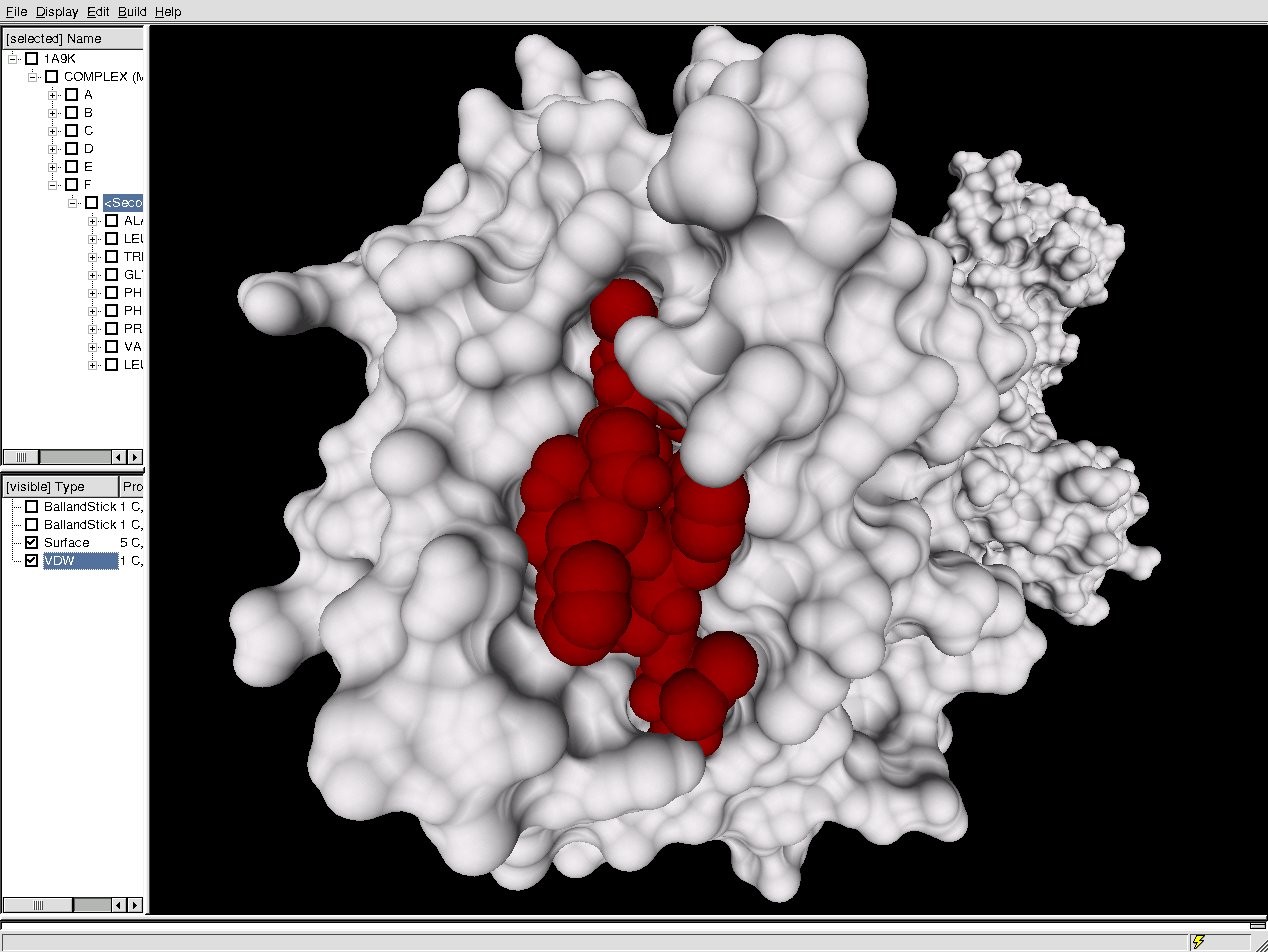
Beispiel eines Protein-Inhibitor-Komplexes in Molview
Teilaufgaben
- GUI (Vorgabe des Zielmoleküls, der Docking-Stelle und des Peptids; Eingabe der Parameter für die Docking-Simulation, Ausgabe der Docking-Resultate)
- Monte-Carlo-Simulation
- Visualisierung der Simulation mittels MolView
- Visualisierung der 3D-Bewegung des Zielmoleküls und des Peptids
- Visualisierung des Energieverlaufs
- Auswertung der Docking-Resultate